<生化学>
遺伝情報発現制御の実行プログラムを理解することは、様々な生命現象の分子メカニズムを解明するために必要です。特に転写活性化・不活性化のperformerである転写調節因子群の機能は、発生・分化、内分泌、脳機能などの高次生命現象のみならず、癌、生活習慣病などの病態にも関わってきます。当研究では、このような概念を根幹に据えて、組織あるいは時期特異的に転写活性を制御する因子、シグナル伝達分子の作用メカニズムなどに関する研究を行っています。
プロモーターやエンハンサーなどの遺伝子発現を調節するDNA領域には、シスエレメントと呼ばれる配列が存在する。シスエレメントとは、転写領域のDNAと同一の分子(すなわちシスの位置)に存在して、転写活性に影響を与える塩基配列のことである。
シスエレメントにはそれに対応するトランスのファクターが存在する。このトランスファクターはタンパク質で、転写因子(転写調節因子、調節タンパク質)と呼ばれる。すなわち、シスエレメントは、特定の転写因子が結合するための目印であり、シスエレメントに転写因子がリクルートされることで転写が調節されるということである。では転写因子は具体的にどのようにして転写活性に影響を与えているのだろうか?これを理解するためには、まず転写について理解しておかなければならない。あまりよく覚えていない人は、また2年次の人体生化学Ⅱの講義内容を復習してほしい。以下、真核細胞における転写、特にmRNAの転写を中心として簡単に説明していきたい。
転写開始点を決定して、転写の頻度を直接的に制御しているゲノム上のDNA配列のことをプロモーターという。最初の方、研究の説明ではプロモーターという言葉を何気なく使ったが、定義としてはそのようなものである。このプロモーター領域にRNAポリメラーゼが結合して転写が始まるが、原核細胞とは異なり、真核細胞のRNAポリメラーゼはプロモーターに直接的に結合することができない。RNAポリメラーゼがプロモーターにリクルートされるためには、基本転写因子と呼ばれるタンパク質の存在が必要である。
プロモーター領域には、いくつかのシスエレメントが存在し、特に重要なのはTATAボックスと呼ばれる配列である。この配列は、転写開始点の上流(5’側)の25塩基対(base pair)付近に存在するチミンとアデニンに富む配列である。このTATAボックスを目印として基本転写因子群がプロモーターに集まり、これを土台にしてRNAポリメラーゼがプロモーターに結合する。その様子を図に示した。ちなみにこの図は人体生化学Ⅱの講義で配布したものに含まれている。もう捨ててしまっただろうか。
図に書かれているTBPとは、TATA Binding Proteinの略で、このタンパク質がTATAボックスに結合する転写因子である。また、TFⅡ(アルファベット:A~H)は、その他の基本転写因子で、TFはTranscription Factorの頭文字である。TFⅡのⅡは、mRNAを合成するRNAポリメラーゼⅡのために使われるものですよ、ということを意味する。アルファベットは便宜上つけられたもので、そのまま名称に使われているが、存在しないもの(例えばTFⅡCとか)もある。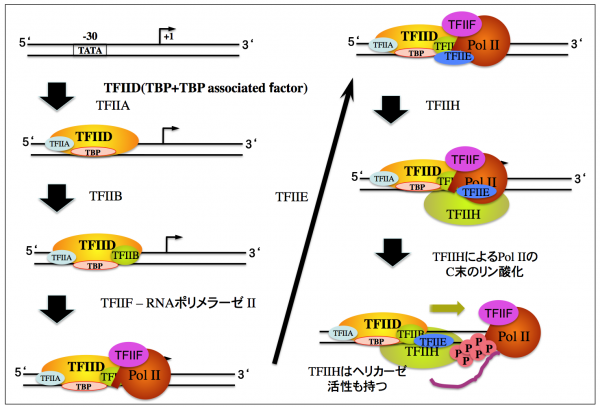
このように、転写の開始を担う重要な機能がプロモーター領域には存在し、特にTATAボックスを中心として基本転写因子やRNAポリメラーゼが集まる領域をコアプロモーターということがある。これに対して、転写開始点から数百塩基対〜数千塩基対以上も離れて、転写活性に影響を及ぼす配列もある。そのような性質を持つもので、転写活性化に関与する領域をエンハンサー、転写抑制に関与する領域をサイレンサーという。これらの領域にもシスエレメントが存在し、そこに特異的に結合する転写因子が存在すると考えられる。このようなエンハンサーやサイレンサーに結合する転写因子には、ある特定の種類の細胞にしか発現しない、いわゆる組織特異的に発現するタンパク質や、外部環境に反応して活性化されるようなタンパク質がある。これら転写因子のはたらきで、生体内では様々な細胞に特有のタンパク質が発現し、それぞれの組織に応じた機能が発揮される。
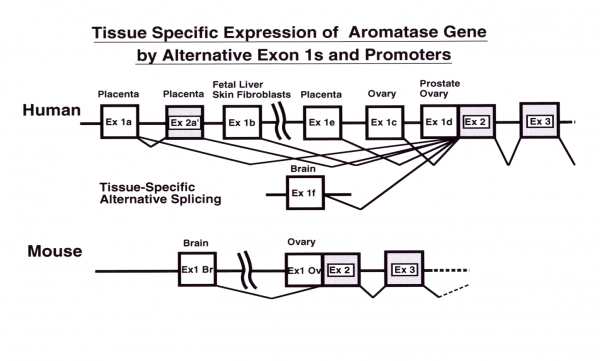
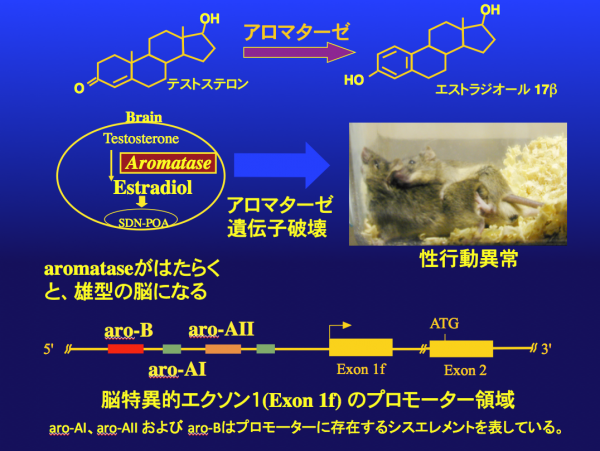
先に挙げたアロマターゼ遺伝子の脳特異的プロモーターに存在するaro-AI, AIIそしてBと名付けた3種のシスエレメントはエンハンサーに分類され、生化学教室ではこれらのシスエレメントに結合する因子を単離し、その性質を調べている。
ここまでの話で、真核生物における転写がどのような仕組みで起こるのか、ある程度イメージしてもらえるようになったとして、転写因子がどのようにして転写活性に影響を与えているのか考えていきたい。転写活性化に関する転写因子の作用としては次のようなことが考えられる。
① 基本転写因子と機能的に相互作用して転写開始複合体の形成を安定化したり、RNAポリメラーゼのリクルートを促進して転写開始を促進する。
② “コアクティベーター”と呼ばれる一群のタンパク質と相互作用してクロマチン構造を変化させる。
③ 転写開始複合体に結合している”メディエーター”と相互作用してRNAポリメラーゼの活性を上昇させる。
アクティベーターとかメディエーターとか別の用語が出てきて混乱しそうですが、それぞれについては調べて勉強してみてください。
研究室の実験内容に戻る。アロマターゼ遺伝子の脳特異的プロモーターには、aro-AI, AIIそしてBと名付けた3種のシスエレメントが存在していると述べたが、これらはどのようにして見つかってきたのか説明したい。まず、プロモーターの性質を調べるためには、プロモーターの活性を測定しないといけない。プロモーターの活性とは、平たく言えば、そのプロモーターがどの程度頻繁に転写を起こすことができるか?ということである。要するに、プロモーターの下流(3’側)に適当なcDNAを連結して(原理的にはタンパクをコードしてなくてもじゃなくても良いのだが・・)、細胞に導入し、転写産物(すなわちRNA)、もしくはそこから翻訳されたタンパク質の量を測れば良い。しかし、RNAやタンパク質の量をそのまま測るのは、なかなか面倒である。RNAを測るにはノーザンブロットや、今話題のRT-PCRを行わないといけない。タンパク質を測るにしてもウエスタンブロットを行う必要があり、どれを取っても時間と手間と費用もかかる。

それで、古くからプロモーター活性を測る時は、活性測定が容易な酵素の遺伝子をプロモーターの下流に置き、発現する酵素活性をもってプロモーター活性とみなすことが多かった。例としてはクロラムフェニコールアセチルトランスフェラーゼ(CAT)とかβガラクトシダーゼとかである。このように、測定しやすい酵素遺伝子を”レポーター”として用いてプロモーター活性を調べる方法を、レポーターアッセイという。現在ではダイナミックレンジが広く、感度も良好なホタルのルシフェラーゼをレポーターに使うことが主流になっている。上の図は、レポーターにCATを使ってアロマターゼ遺伝子の脳特異的プロモーター活性を調べた結果を示している。プロモーター領域を上流約950塩基対のところから、少しづつ短くしていくと、プロモーター活性がところどころ変化していることがわかる。特に上流約200塩基対(-202)まで削り込んだところで、活性が高くなっている。さらに約80塩基対(-82)まで削り込むと、活性が極端に落ち込んでいることから、200から80塩基対の間に重要なシスエレメントが存在することがわかる。-418から-202塩基対まで切り込んだ場合は、活性がむしろ増加しているが、おそらくこの領域にサイレンサーが存在しているためであろう。
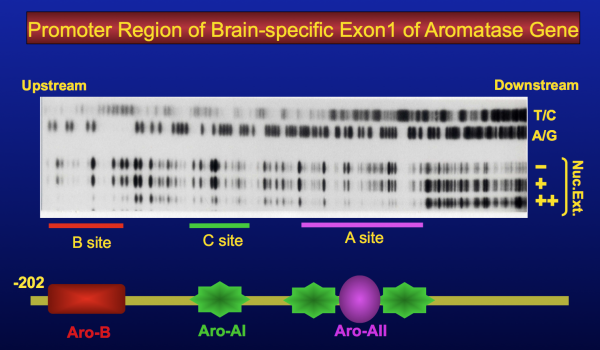
シスエレメントの位置がある程度絞り込めたが、まだまだ領域が広い。転写因子の認識するシスエレメント(塩基配列)の長さは、せいぜい6塩基対、長くても10塩基対程度なので、もっと狭まるはずである。そこでフットプリントアッセイという方法を使って、シスエレメントの詳細な位置を調べることにした。DNAに核抽出液を混ぜると、その中に含まれる転写因子がDNAの特異的な配列に結合する。この状態でDNA分解酵素を弱い条件で作用させると、タンパク質が結合していないDNAの部分は、ランダムな位置で分解を受ける。そしてポリアクリルアミドゲル電気泳動を利用して鎖長にしたがって分離すると、はしご状のパターンとなる。この時、タンパク質(転写因子)が結合している部分はDNAの分解から保護されるために切断されず、結果的にその部分が、はしご状のバンドの空白部分として検出されるというものである(上図)。
最終的には、ゲルシフトアッセイなどを組み合わせることにより、先に述べた3つのシスエレメントを同定できた。
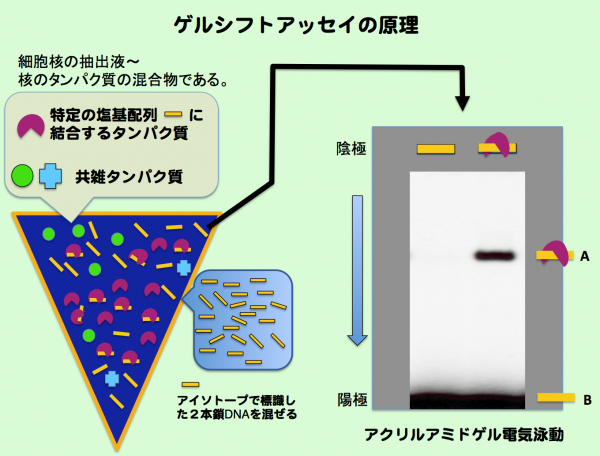
ゲルシフトアッセイの原理についても説明しておきたい。なんだか難しそうな名前だが、細胞や組織から核の抽出液を調製して、放射能ラベルした短い、20塩基対くらいの合成DNAと混ぜ、電気泳動するだけである(上図)。合成DNAはその名の通り、人工的に使ったDNAだが、その塩基配列は、先のシスエレメントを含む部分を使う。このDNAをゲルシフトにおけるプローブと呼ぶ。核抽出液にプローブを混ぜると、抽出液に含まれるDNA結合タンパク質(転写因子)がプローブ中のシスエレメントと、”特異的に”結合する。この反応液を電気泳動すると、タンパク質と結合していないプローブは図のBの位置まで移動しているのに対し、タンパク質と結合したDNAはその分大きくなるので移動度が小さく、Aの位置までしか移動しない。ゲルシフトアッセイは、その名が示す通り、ゲル中での移動度がシフトするのを測ることで、タンパク質— DNA複合体の形成を調べる方法である。
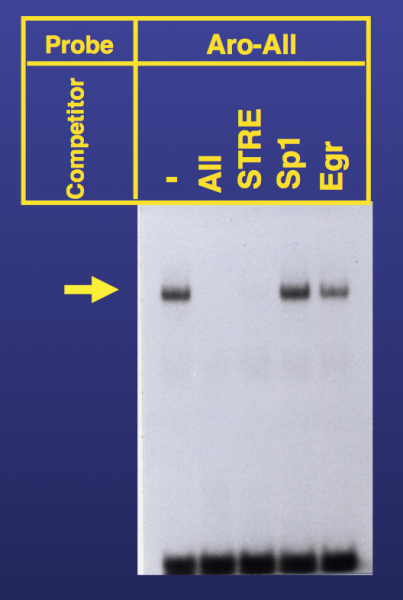
実際の例を見てみよう、上図はアロマターゼプロモーターのaro-AⅡサイトを含むDNAをプローブとして用いたゲルシフトアッセイの結果である。この図の中でプローブの他に、コンペティター(competitor)というものを使っているのに気が付くだろう。ここに示したコンペティターとは、放射性ラベルをしていないプローブのことである。すなわち、放射能活性を持たないプローブを大量に(100~200倍くらい)反応液に加えると、放射性のプローブが追い出される、まさにコンペティション(競合)が生じて、シフトしたバンドが消失してしまう現象が起こる。酵素阻害のところで学んだ「競合阻害」を思い出してもらうとすんなり理解できると思う。ゲルシフトアッセイでは、DNAならどんな塩基配列でも競合が起きるようであれば、それは”特異的に”結合していないということになる。タンパク質とDNAの結合が特異的でなければ、生理的意味があやふやになるのである。図の中のSTRE、Sp1、そしてEgrはいずれも異なるシスエレメントである。放射性ラベルをしていないaro-AⅡや、これらのプローブをコンペティターとして反応液に加えてみる。aro-AⅡをコンペティターとしたレーンでは、矢印で示したバンドが消失している。これは放射性プローブと同じものを競合させているので当然である。この時、実際にはタンパク質— DNA複合体の形成は生じてはいるが、複合体中のプローブが、ラベルされていないプローブと置き換わったので(オートラジオグラムで)検出できなくなっただけであることに注意しよう。そのほかのシスエレメントについて見てみると、STREでも競合が起きているが、Sp1やEgrといったシスエレメントでは競合が起きていない(すなわちバンドが消えていない)。このことより、aro-AⅡに結合するタンパク質はSTREに結合するものと同じであるが、Sp1やEgrに結合するものとは異なると推測できる。
もう一つ応用例を見てみよう。下の図はゲルシフトアッセイにおける様々なプローブの競合を見たものである。コンペティターとして用いたプローブは、元のaro-AⅡプローブと、aro-AⅡの塩基配列に連続的な変異を導入したものを使用している。図は塩基配列と比べやすいように、バンドの部分だけを抜き出して縦に示している。変異した部分は、それぞれの変異体(AⅡM1〜AⅡM9)の中で示し、元のプローブと同じ塩基は、ハイフン(—)で表している。図を見てわかるように、赤字で示した塩基を変異したプローブでは競合が起きなくなる(バンドが消えない)ことから、この部分がタンパク質との結合における必須の塩基配列であることがわかる。言い換えると、aro-AⅡ結合タンパク質は、この3′-TTGGCCCCT-5′ 配列で示されるシスエレメントに特異的に結合して、その機能を発揮しているということである。

次に気になるのは、このaro-AⅡ結合タンパク質とは、どんなタンパク質なのかということ、”aro-AⅡ結合タンパク質”の正体である。
ここで、みんなに質問したい。もしあなたなら、このaro-AⅡ結合タンパク質を捕まえる(単離する)ために、どのような方法をとるだろうか?
今までに学んだ、機能形態学、生体機能分子学、人体生化学、人体機能調節学、分析化学などの知識を総動員して考えて見て欲しい。
ただし、この問いに明確な正解は無い、というか無数にあるとも言えるので、自由に考えてください。
もし良い考えがまとまったら、こちらまで(sihonda@fukuoka-u.ac.jp).
ある方法により、なんとかaro-AⅡ結合タンパク質のcDNAクローンを単離できた。このcDNAにはARP1という核内受容体ファミリーの一つがコードされていた。次に単離したARP1が、本当にaro-AⅡ配列に結合するのかってことを確かめなければいけない。aro-AⅡ結合を指標に採ってきたんだから結合するのは当たり前だろう・・という一般人からすれば常識のような考え方は通用しない。すでにcDNAが取れたので、確認するのはある程度簡単である。ARP1のcDNAを適当なプラスミドベクターにサブクローンして大腸菌で増やして精製した。今回使用したのは、pCI-neoというベクターで、T7ファージのプロモーターを持っていて、in vitro転写でmRNA合成ができる。合成したmRNAをウサギの網状赤血球ライセートに加えると、加えたmRNAに基づいてタンパク質が合成される。このようにin vitroで合成したARP1タンパク質を用いて、先のゲルシフトアッセイを行なった結果がこの図である。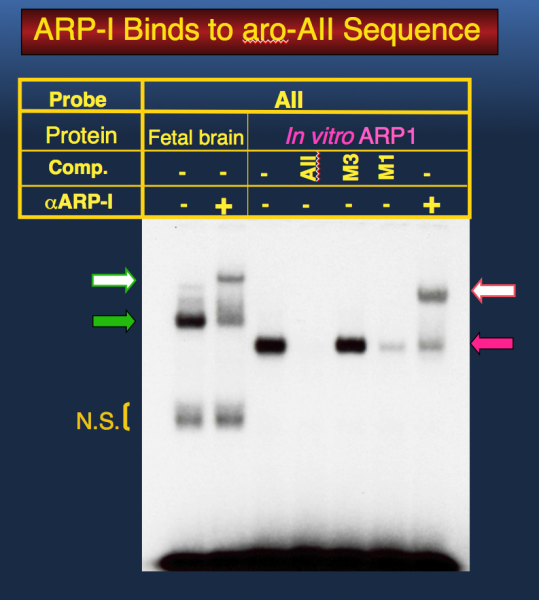 Fetal brainと書いたのが胎仔の脳から調製した核抽出液を用いたもの(緑の矢印)、In Vitro ARP1と書いたのがin vitro合成ARP1を用いたもの(赤の矢印)で、in vitro合成ARP1の方が若干小さい複合体を形成しているようである。実は単離できたARP1のクローンはアミノ末端の9アミノ酸が欠けていて、それをそのままこの実験に使ったものだから、その分小さいタンパク質ができているのではないかと考えているが、これは推測であり、この移動度の違いが欠けた9アミノ酸のせいなのかどうかは実際には不明である。full length(全長)のクローンをとって確認すればいいのだが、確認はしていない。もしかすると生体内でのARP1は、何かしらの翻訳後修飾を受けて分子量が大きくなっているのかもしれない。 また、白抜きになっている矢印は抗ARP1抗体を反応液に加えた時に、より移動度が小さくなるシグナルを指している。これは複合体の中にARP1タンパク質が含まれていると、そこに抗体が結合してさらに大きな複合体となり、結果的に移動度が小さくなるものと解釈できる。すなわち、ある抗体を加えて、このように元のシグナルよりも移動度の小さいシグナル(スーパーシフトという)が認められれば、その複合体の中には、加えた抗体に対する抗原が含まれているということである。このようにIn Vitro で合成したARP1は核抽出液を用いた時と同じ結合特性を示し、胎仔脳から調製した核抽出液に含まれていてaro-AⅡ結合に結合するタンパク質はARP1であることが証明できた。
Fetal brainと書いたのが胎仔の脳から調製した核抽出液を用いたもの(緑の矢印)、In Vitro ARP1と書いたのがin vitro合成ARP1を用いたもの(赤の矢印)で、in vitro合成ARP1の方が若干小さい複合体を形成しているようである。実は単離できたARP1のクローンはアミノ末端の9アミノ酸が欠けていて、それをそのままこの実験に使ったものだから、その分小さいタンパク質ができているのではないかと考えているが、これは推測であり、この移動度の違いが欠けた9アミノ酸のせいなのかどうかは実際には不明である。full length(全長)のクローンをとって確認すればいいのだが、確認はしていない。もしかすると生体内でのARP1は、何かしらの翻訳後修飾を受けて分子量が大きくなっているのかもしれない。 また、白抜きになっている矢印は抗ARP1抗体を反応液に加えた時に、より移動度が小さくなるシグナルを指している。これは複合体の中にARP1タンパク質が含まれていると、そこに抗体が結合してさらに大きな複合体となり、結果的に移動度が小さくなるものと解釈できる。すなわち、ある抗体を加えて、このように元のシグナルよりも移動度の小さいシグナル(スーパーシフトという)が認められれば、その複合体の中には、加えた抗体に対する抗原が含まれているということである。このようにIn Vitro で合成したARP1は核抽出液を用いた時と同じ結合特性を示し、胎仔脳から調製した核抽出液に含まれていてaro-AⅡ結合に結合するタンパク質はARP1であることが証明できた。
次の問題は、ARP1の機能についてである。先に説明したレポーターアッセイを応用して、ARP1のアロマターゼ脳特異的プロモーターに及ぼす影響を調べたのがこの下の図である。図に示した様にルシフェラーゼ構造遺伝子に脳特異的プロモーター(上流200 bps)を含んだ領域を連結したレポータープラスミド(pGL3aroBr)を構築した。これを内因性にARP1を発現していないCV1細胞にトランスフェクトすると、プロモーター活性に従ってルシフェラーゼが発現するが、pGL3aroBrだけでは、プロモーター配列を持っていないプラスミド(pGL3)と同程度の活性しかない。ここでARP1の発現プラスミドを同時にトランスフェクト(コ-トランスフェクションという)すると、ARP1発現プラスミドの量を増やすにしたがって、すなわち用量依存的に、プロモーター活性が上昇していることがわかる。考え方としては、ARP1発現プラスミドとpGL3aroBrをコトランスフェクションすると、まずARP1が発現してタンパク質ができる。そしてそのARP1がpGL3aroBrのaroAIIサイトに結合して、ルシフェラーゼ遺伝子の転写を促進しているということである。ARP1発現プラスミドの量を増やすと、それだけARP1タンパク質の量も増えるので、より活性化が大きくなると考えられる。この様に、ARP1はアロマターゼ脳特異的プロモーターに存在するaro-AIIサイトに結合し、転写を正に制御するようにはたらいているようである。

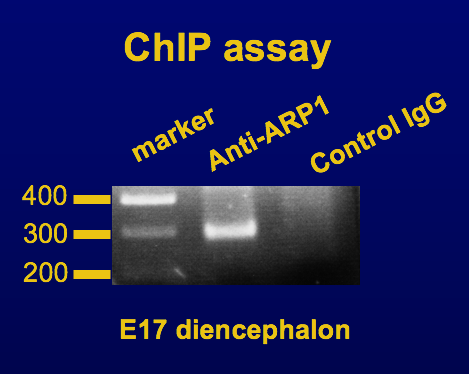
次にマウスの胎仔の脳で、ARP1が同様な機能をしているかどうか調べた。脳の中でARP1がAroAIIサイトに結合しているかどうか調べるため、クロマチン免疫沈降法(ChIP法)を行った。ChIP法は、次の手順で行われる。まず、生きた培養細胞や組織などをホルムアルデヒドで処理し、DNAとそれに結合しているタンパク質を固定化する。その後、超音波処理などでDNAを500 bp以下に断片化し、この試料に対して目的のタンパク質(今回 ARP1)の抗体を用いて免疫沈降を行う。そしてタンパク質と共に沈殿したDNA領域をエンドポイントPCRやqRT-PCRによって検出する。この方法で、検出したDNA領域には目的のタンパク質が結合していると判断できる。今回、胎生17日目(E17)の間脳部を用いてChIPを行うと、下図に示すように抗ARP1抗体で免疫沈降した場合にのみ、AroAIIサイト付近のDNAが検出されることがわかった。このことより、in vivoにおいてもARP1はAroAIIサイトに結合することが明らかとなった。
次にARP1が脳特異的プロモーターの活性に及ぼす影響も調べた。株化された培養細胞で脳特異的プロモーターを利用してアロマターゼを発現するものはないが、胎仔から間脳部を取り出して初代培養するとシャーレの中でもアロマターゼを発現することがわかっている。しかも、胎生13日目の脳から初代培養細胞を調製すると、生体の時と同じように、培養4日目まで、アロマターゼmRNA量が増加する。この実験系を利用して、ARP1をsiRNAによってノックダウンすると、胎仔脳初代培養細胞におけるアロマターゼの発言がどうなるかを調べたのが下図である。
WBはウエスタンブロットの結果で、ARP1のsiRNAを導入するとARP1タンパク質がノックダウンされ、その量が著しく減少していることがわかる。
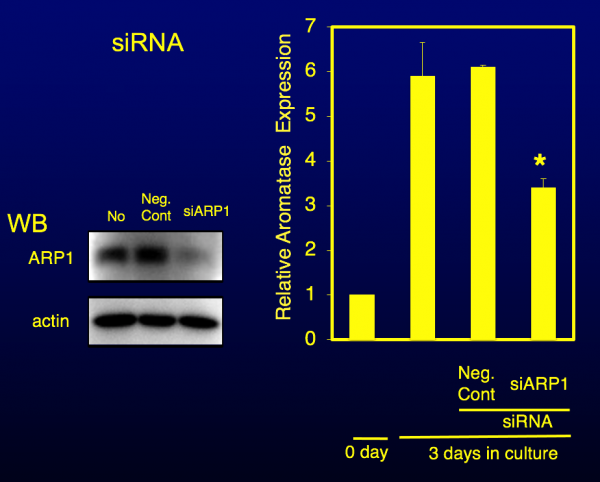
その右隣のグラフは、アロマターゼmRNAをqRT-PCRで測定したものである。0dayは初代培養に移した初日のもので、3日後(3 days)には、その量が6倍近くに増えているのがわかる。ところがARP1をノックダウンすると、その発現量はネガティブコントロールに比べて半分ほどに低下している。このように実際に生体内でもARP1はアロマターゼの脳特異的プロモーターに結合し、その転写活性を正に制御していることが明らかとなった。
今回の実験において、ARP1が脳のアロマターゼの遺伝子発現に関与することがわかったが、脳特異的プロモーターに結合する転写因子は他にも存在する。ARP1がなくても完全に転写活性が落ちてしまわないのは、そのほかのシスエレメント、Aro-BやAro-AIなどニケツ合意する転写因子の存在が、転写を助けているからかもしれない。今後は、これらのシスエレメントに結合する転写因子について調べることが必要である。また、DNAに直接結合する転写因子は、さらに転写共役因子をリクルートすることが知られている。そのような転写共役因子は、ヒストンアセチルトランスフェラーゼやヒストンデアセチラーゼなどの酵素活性を有するものが多く、その活性により転写を促進したり抑制したりしている。今後、ARP1に結合する因子を単離してその性質を調べ、本遺伝子の転写制御メカニズムを総合的に理解することが重要である。
今回紹介したARP1などの転写因子による遺伝子発現制御機構は、アロマターゼ遺伝子のためにだけに用意されているとは考えにくい。ARP1は胎仔脳のいろいろな領域の神経細胞に発現していることから、アロマターゼ以外の遺伝子の発現にも関与していることは容易に推測できる。したがって、このような転写因子の機能メカニズムを調べることは、アロマターゼのような特殊な遺伝子から、中枢神経系で発現する普遍的な遺伝子の発現制御機構への理解、さらには脳の性分化など高次脳機能形成のメカニズムを理解することに繋がると考えている。